Vad är GLM i R?
Generaliserade linjära modeller är en delmängd av linjära regressionsmodeller och stöder icke-normala distributioner effektivt. För att stödja detta rekommenderas att du använder glm () -funktionen. GLM fungerar bra med en variabel när variansen inte är konstant och distribueras normalt. En länkfunktion definieras för att transformera svarsvariabeln så att den passar till lämplig modell. En LM-modell görs med både familjen och formeln. GLM-modellen har tre viktiga komponenter som kallas slumpmässig (sannolikhet), systematisk (linjär prediktor), länkkomponent (för logit-funktion). Fördelen med att använda glm är att de har modellflexibilitet, inget behov av konstant varians och den här modellen passar maximal sannolikhetsberäkning och dess förhållanden. I det här ämnet kommer vi att lära oss om GLM i R.
GLM-funktion
Syntax: glm (formel, familj, data, vikter, delmängd, Start = null, modell = SANT, metod = ”” …)
Här inkluderar familjetyper (inkluderar modelltyper) binomial, poisson, gaussisk, gamma, kvasi. Varje distribution utför en annan användning och kan användas i antingen klassificering och förutsägelse. Och när modellen är gaussisk, bör svaret vara ett riktigt heltal.
Och när modellen är binomial bör svaret vara klasser med binära värden.
Och när modellen är Poisson, bör svaret vara icke-negativt med ett numeriskt värde.
Och när modellen är gamma, bör svaret vara ett positivt numeriskt värde.
glm.fit () - För att passa en modell
Lrfit () - anger logistisk regressionspassning.
uppdatering () - hjälper till att uppdatera en modell.
anova () - det är ett valfritt test.
Hur man skapar GLM i R?
Här ska vi se hur man skapar en lätt generaliserad linjär modell med binära data med glm () -funktion. Och genom att fortsätta med Trees-datauppsättningen.
exempel
// Importera ett biblioteklibrary(dplyr)
glimpse(trees)

För att se kategoriska värden tilldelas faktorer.
levels(factor(trees$Girth))
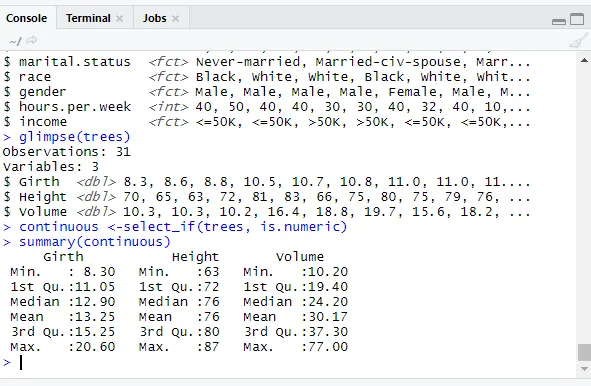
// Verifiera kontinuerliga variabler
library(dplyr)
continuous <-select_if(trees, is.numeric)
summary(continuous)
// Inklusive träddatasats i R-sökning Pathattach (träd)
x<-glm(Volume~Height+Girth)
x
Produktion:
| Samtal: glm (formel = Volym ~ Höjd + Omkrets)
koefficienter: (Intercept) Höjdomkrets -57, 9877 0, 3393 4, 7082 Degrees of Freedom: 30 Total (dvs. Null); 28 Rest Null avvikelse: 8106 Restavvikelse: 421.9 AIC: 176.9 |
summary(x)
| Ring upp:
glm (formel = Volym ~ Höjd + Omkrets) Avvikelse rester: Min 1Q Median 3Q Max -6.4065 -2.6493 -0.2876 2.2003 8.4847 koefficienter: Uppskatta Std. Fel t-värde Pr (> | t |) (Intercept) -57.9877 8.6382 -6.713 2.75e-07 *** Höjd 0.3393 0.1302 2.607 0.0145 * Omkrets 4.7082 0.2643 17.816 <2e-16 *** - Signif. koder: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0, 1 '' 1 (Dispersionsparameter för gaussisk familj anses vara 15.06862) Noll avvikelse: 8106.08 på 30 frihetsgrader Restavvikelse: 421, 92 på 28 frihetsgrader AIC: 176, 91 Antal iterationer för Fisher-poäng: 2 |
Utgången från sammanfattningsfunktionen ger upp samtal, koefficienter och rester. Ovanstående svar visar att både höjd och omkrets är effektiva utan betydelse eftersom sannolikheten för dem är mindre än 0, 5. Och det finns två varianter av avvikelse som heter null och resterande. Slutligen är fisherscoring en algoritm som löser maximala sannolikhetsproblem. Med binomial är svaret en vektor eller matris. cbind () används för att binda kolumnvektorerna i en matris. Och för att få detaljerad information om fit-sammanfattningen används.
För att göra som huva-test körs följande kod.
step(x, test="LRT")
Start: AIC=176.91
Volume ~ Height + Girth
Df Deviance AIC scaled dev. Pr(>Chi)
421.9 176.91
- Height 1 524.3 181.65 6.735 0.009455 **
- Girth 1 5204.9 252.80 77.889 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Call: glm(formula = Volume ~ Height + Girth)
Coefficients:
(Intercept) Height Girth
-57.9877 0.3393 4.7082
Degrees of Freedom: 30 Total (ie Null); 28 Residual
Null Deviance: 8106
Residual Deviance: 421.9 AIC: 176.9
Modell passform
a<-cbind(Height, Girth - Height)
> a
sammanfattning (träd)
Girth Height Volume
Min. : 8.30 Min. :63 Min. :10.20
1st Qu.:11.05 1st Qu.:72 1st Qu.:19.40
Median :12.90 Median :76 Median :24.20
Mean :13.25 Mean :76 Mean :30.17
3rd Qu.:15.25 3rd Qu.:80 3rd Qu.:37.30
Max. :20.60 Max. :87 Max. :77.00
För att få lämplig standardavvikelse
apply(trees, sd)
Girth Height Volume
3.138139 6.371813 16.437846
predict <- predict(logit, data_test, type = 'response')
Därefter hänvisar vi till räknaresponsvariabeln för att modellera en bra responspassning. För att beräkna detta kommer vi att använda USAccDeath-datasättet.
Låt oss ange följande utdrag i R-konsolen och se hur årräkningen och årstorget utförs på dem.
data("USAccDeaths")
force(USAccDeaths)
// För att analysera året 1973-1978.
disc <- data.frame(count=as.numeric(USAccDeaths), year=seq(0, (length(USAccDeaths)-1), 1)))
yearSqr=disc$year^2
a1 <- glm(count~year+yearSqr, family="poisson", data=disc)
summary(a1)
| Ring upp:
glm (formel = räkning ~ år + årSqr, familj = “poisson”, data = skiva) Avvikelse rester: Min 1Q Median 3Q Max -22.4344 -6.4401 -0.0981 6.0508 21.4578 koefficienter: Uppskatta Std. Fel z-värde Pr (> | z |) (Intercept) 9.187e + 00 3.557e-03 2582.49 <2e-16 *** år -7.207e-03 2.354e-04 -30.62 <2e-16 *** årSkr 8.841e-05 3.221e-06 27.45 <2e-16 *** - Signif. koder: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0, 1 '' 1 (Dispersionsparameter för Poisson-familjen anses vara 1) Noll avvikelse: 7357, 4 på 71 frihetsgrader Restavvikelse: 6358, 0 på 69 frihetsgrader AIC: 7149, 8 Antal iterationer för Fisher-poäng: 4 |
För att kontrollera att modellen passar bäst kan följande kommando användas för att hitta
resterna för testet. Från nedanstående resultat är värdet 0.
1 - pchisq(deviance(a1), df.residual(a1))
Använda QuasiPoisson-familjen för att få större variationer i de givna uppgifterna
a2 <- glm(count~year+yearSqr, family="quasipoisson", data=disc)
summary(a2)
| Ring upp:
glm (formel = räkna ~ år + årSqr, familj = “quasipoisson”, data = skiva) Avvikelse rester: Min 1Q Median 3Q Max -22.4344 -6.4401 -0.0981 6.0508 21.4578 koefficienter: Uppskatta Std. Fel t-värde Pr (> | t |) (Intercept) 9.187e + 00 3.417e-02 268.822 <2e-16 *** år -7.207e-03 2.261e-03 -3.188 0.00216 ** årSqr 8.841e-05 3.095e-05 2.857 0.00565 ** - (Dispersionsparameter för quasipoisson-familjen anses vara 92.28857) Noll avvikelse: 7357, 4 på 71 frihetsgrader Restavvikelse: 6358, 0 på 69 frihetsgrader AIC: NA Antal iterationer för Fisher-poäng: 4 |
Att jämföra Poisson med binomialt AIC-värde skiljer sig avsevärt. De kan analyseras med precision och återkallningsförhållande. Nästa steg är att verifiera rester avvikelse är proportionell mot medelvärdet. Då kan vi plotta med ROCR-bibliotek för att förbättra modellen.
Slutsats
Därför har vi fokuserat på en speciell modell som kallas generaliserad linjär modell som hjälper till att fokusera och uppskatta modellparametrarna. Det är främst potentialen för en kontinuerlig responsvariabel. Och vi har sett hur glm passar R-inbyggda paket. De är de mest populära metoderna för att mäta räknedata och ett robust verktyg för klassificeringstekniker som används av en datavetare. R-språk hjälper naturligtvis till att göra komplicerade matematiska funktioner
Rekommenderade artiklar
Detta är en guide till GLM i R. Här diskuterar vi GLM-funktionen och hur man skapar GLM i R med träddatainställningar exempel och utdata. Du kan också titta på följande artikel för att lära dig mer -
- R Programmeringsspråk
- Big Data Arkitektur
- Logistisk regression i R
- Big Data Analytics-jobb
- Poisson Regression i R | Implementering av Poisson Regression